Menu dodatkowe
Menu strony
Ścieżka nawigacyjna
Ikony społecznościowe
Treść strony
Identyfikacja molekularna nicieni
Diagnostyczne metody molekularne stosowane do identyfikacji kwarantannowego nicienia węgorka sosnowca (Bursaphelenchus xylophilus)
Wstęp
Skuteczne ograniczanie rozprzestrzeniania kwarantannowego nicienia węgorka sosnowca (B. xylophilus) na drodze eliminacji zainfekowanego materiału drzewnego jest niemożliwe bez precyzyjnej identyfikacji występujących w drewnie nicieni. Nicienie należące do rodzaju Bursaphelenchus są organizmami morfologicznie bardzo podobnymi do siebie, dlatego przy ich ekstrakcji z prób drewna konieczne jest stosowanie metod identyfikacyjnych gwarantujących jak najbardziej precyzyjne ich rozpoznanie. Szybka metoda identyfikacji szkodnika na podstawie jego cech morfologicznych może być zawodna, jeśli nie jest przeprowadzana przez specjalistę nematologa mającego dostęp do szerokiego materiału referencyjnego. W przypadku międzynarodowego transportu surowca drzewnego równie ważnym kryterium o znaczeniu ekonomicznym jest możliwie najkrótszy czas przeprowadzania takich analiz, ponieważ prawidłowe odróżnienie B. xylophilus od pozostałych, nieszkodliwych gatunków może również zaoszczędzić znacznych nakładów na podejmowanie nieuzasadnionych interwencji.
Zmienność morfologiczna nicieni z rodzaju Bursaphelenchus należących do grupy xylophilus (szczególnie trzech gatunków z tej grupy naturalnie występujących w Europie, tj. B. mucronatus, B. fraudulentus i B. populi) stanowi podstawowe utrudnienie w ostatecznej identyfikacji taksonomicznej tych gatunków wyłącznie na podstawie cech morfologicznych. Wobec zaistniałej sytuacji szczególnego znaczenia nabrały badania nad identyfikacją taksonomiczną nicieni z rodzaju Bursaphelenchus oraz doskonaleniem precyzji i szybkości metod dostępnych dla służb kwarantannowych. Precyzja identyfikacji taksonomicznej nicieni w znacznym stopniu polepszona została dzięki wykorzystaniu metod biologii molekularnej, co okazało się istotnie ważne w przypadku identyfikacji nicieni z grupy ‘xylophilus’. Doprowadziło to jednocześnie to wykrycia wielu nowych gatunków z rodzaju Bursaphelenchus.
Poniżej opisane zostały rekomendowane przez nasze laboratorium techniki molekularne wykorzystywane do identyfikacji kwarantannowego nicienia węgorka sosnowca.
1. PCR–RFLP
Stosowanie techniki PCR–RFLP stało się w ostatnich latach częścią standardowego zestawu metod identyfikacji nicieni z rodzaju Bursaphelenchus, należących do protokołu diagnostycznego kwarantannowego nicienia B. xylophilus (EPPO Bulletin, 2013) (Rys. 13.) Zastosowane dla badanych gatunków uniwersalne startery amplifikują regiony ITS wszystkich nicieni z rodzaju Bursaphelenchus powodując uzyskiwanie produktów PCR wielkości od 930 do 1030 pz. W analizie restrykcyjnej stosuje się pięć enzymów restrykcyjnych (RsaI, HaeIII, MspI, HinfI i AluI), których skuteczność została wcześniej wykazana przy identyfikacji różnych gatunków nicieni z rodzaju Bursaphelenchus (Burgermeister i in., 2009). W naszych badaniach metoda ta wykorzystywana jest do weryfikacji taksonomicznej i identyfikacji wszystkich, wykrytych w Polsce gatunków z rodzaju Bursaphelenchus, które morfologicznie zbliżone są do grupy xylophilus, w tym do węgorka sosnowca. Opierając się na porównaniach znanych dotychczas profili restrykcyjnych z profilami uzyskanymi w naszych badaniach prowadzonych na licznych populacjach Bursaphelenchus spp., izolowanych z różnych obszarów Polski, potwierdzona została przynależność części tych populacji do gatunków B. mucronatus, B. fraudulentus, lub do nowego B. populi, należących do grupy ‘xylophilus’ (Tomalak i Filipiak, 2010) (Ryc. 14-16). W ostatniej dekadzie opisaliśmy również serię innych, nowych gatunków nicieni z tego rodzaju, tj. B. trypophloei, B. masseyi, B. fagi, B. tiliae, B. piceae, B. taphrorychi, i B. michalskii, wykorzystując przy tym technikę PCR-RFLP. Wszystkie profile uzyskane dla tych nicieni istotnie różniły się od profili uzyskanych dla różnych populacji kwarantannowego nicienia B. xylophilus (Ryc. 13).
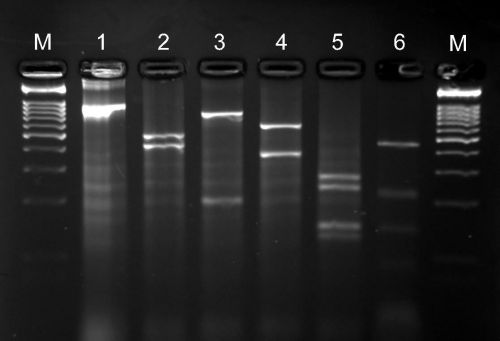
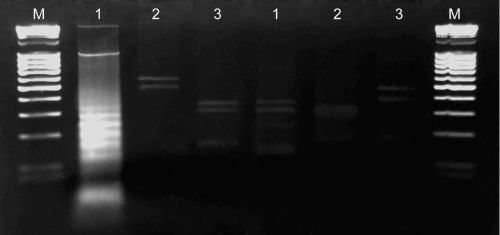
Ryc. 13. Profile PCR–RFLP gatunku Bursaphelenchus xylophilus: 1 – produkt reakcji PCR,
2–6 – produkty PCR trawione enzymami restrykcyjnymi: 2 – RsaI, 3 – HaeIII, 4 – MspI,
5 – HinfI, 6 – AluI, M – marker DNA (MassRuler™, Fermentas)
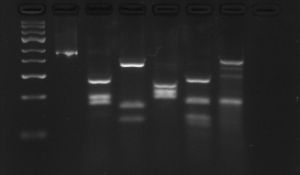
Ryc. 14. Profile PCR–RFLP odrębnych polskich izolatów gatunku Bursaphelenchus mucronatus prezentujących różne genotypy, odpowiednio genotyp europejski, wschodnio-azjatycki i pośredni. : 1 – marker; 2 – produkt reakcji PCR, 3–7 – produkty PCR trawione enzymami restrykcyjnymi : 3 – RsaI, 4 – HaeIII, 5 – MspI, 5 – HinfI, 6 – AluI, M – marker DNA (MassRuler™, Fermentas)
Ryc. 14.1. Profile PCR–RFLP gatunku Bursaphelenchus mucronatus ssp. kolymensis (genotyp europejski: Olsztyn-01).
Ryc. 14.2. Profile PCR–RFLP gatunku Bursaphelenchus mucronatus ssp. mucronatus (genotyp wschodnio-azjatycki: Kampinos-01).
Ryc. 14.3. Profile PCR–RFLP gatunku Bursaphelenchus mucronatus, prezentujące genotyp pośredni (Lwówek-01).
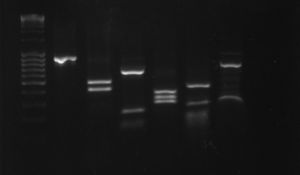
Ryc. 15. Profile PCR–RFLP gatunku Bursaphelenchus fraudulentus: 1 – produkt reakcji PCR,
2–6 – produkty PCR trawione enzymami restrykcyjnymi: 2 – RsaI, 3 – HaeIII, 4 – MspI,
5 – HinfI, 6 – AluI, M – marker DNA (MassRuler™, Fermentas)
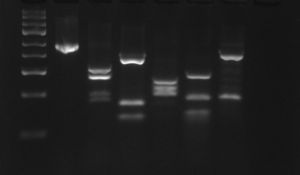
Ryc. 16. Profile PCR–RFLP gatunku Bursaphelenchus populi: 1 – produkt reakcji PCR,
2–6 – produkty PCR trawione enzymami restrykcyjnymi: 2 – RsaI, 3 – HaeIII, 4 – MspI,
5 – HinfI, 6 – AluI, M – marker DNA (MassRuler™, Fermentas)
2. Real-time PCR–HRM
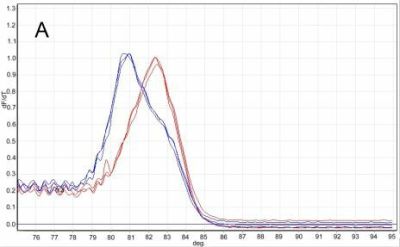
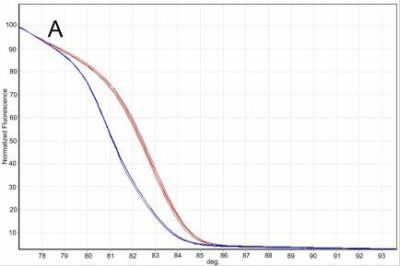
Technika real-time PCR–HRM została w naszych badaniach po raz pierwszy opracowana i wykorzystana do wykrywania kwarantannowego szkodnika węgorka sosnowca. Dużą zaletą opracowanej metody jest skuteczność odróżnienia tego nicienia od populacji B. mucronatus, należących do obu znanych podgatunków (tj. prezentujących genotyp europejski, lub wschodnio-azjatycki). W wyniku przeprowadzonej analizy, otrzymywane były wyraźnie różne krzywe topnienia produktów reakcji dla węgorka sosnowca (82.44°C) oraz wyraźnie innego produktu dla populacji B. mucronatus należących do genotypu europejskiego (80.74°C) i dwóch produktów amplifikacji dla populacji B. mucronatus należących do genotypu wschodnio-azjatyckiego (80.74°C i 82.34°C), które w łatwy sposób pozwalały odróżnić od siebie te dwa gatunki nicieni (Ryc. 17.) (szczegóły metodyczne podane są w publikacji Filipiak i Hasiów-Jaroszewska, 2016).
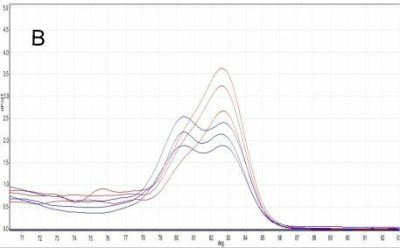
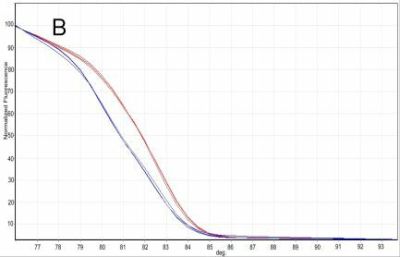
W wyniku serii 10-krotnych rozcieńczeń wykazano, że stężenie 500 fg/μl było najniższą koncentracją DNA wykrywaną w opracowanej technice. Uzyskane podczas analizy HRM znormalizowane krzywe topnienia DNA różniły się pomiędzy sobą temperaturą denaturacji, o czym świadczyły znaczne przesunięcia tych krzywych względem siebie (Ryc. 18). Wskazuje to na obecność różnic w składzie nukleotydowym badanych gatunków nicieni w amplifikowanym rejonie genomu. Przeprowadzone badania wykazały ponadto, że identyczne wyniki można uzyskać zarówno na podstawie DNA izolowanego z osobników dorosłych, jak i ze stadiów larwalnych. Prace te potwierdziły, że dzięki wykrywaniu pojedynczych zmian nukleotydowych w badanych produktach PCR, technika real-time PCR–HRM jest bardzo przydatną metodą do wykrywania kwarantannowego nicienia węgorka sosnowca.
Ryc. 17. Krzywe topnienia reakcji real-time PCR-HRM dla B. xylophilus (kolor czerwony) i B. mucronatus (kolor niebieski) dla genotypu europejskiego (A) i wschodnio-azjatyckiego (B).
Ryc. 18. Znormalizowany wykres fluorescencji różnicujący produkty reakcji real-time PCR-HRM dla B. xylophilus (kolor czerwony) i B. mucronatus (kolor niebieski) dla genotypu europejskiego (A) i wschodnio-azjatyckiego (B).
3. Molekularne metody do jednoczesnego wykrywania i identyfikacji kwarantannowego nicienia B. xylophilus oraz niepatogenicznych B. mucronatus i B. fraudulentus
Z uwagi na bliskie pokrewieństwo morfologiczne kwarantannowego nicienia węgorka sosnowca i dwóch innych, rodzimych dla Europy niepatogenicznych gatunków z grupy ‘xylophilus’, tj. B. mucronatus i B. fraudulentus, i w związku z tym możliwość błędnej ich identyfikacji w próbach drewna sosny, opracowane zostały metody molekularne umożliwiające jednoczesne wykrywanie i identyfikację tych trzech gatunków nicieni. W przypadku kolonizacji nowych obszarów przez B. xylophilus nicień ten może zasiedlać te same drzewostany i drzewa sosny oraz chrząszcze żerdzianki sosnówki co powszechny w Europie B. mucronatus. Może on również zasiedlać te same drzewa, co B. fraudulentus, który związany jest zwykle z drzewami liściastymi, lecz okazyjnie notowany jest w Polsce również w drzewach iglastych, m.in. w sośnie i modrzewiu.
W naszych badaniach opracowane zostały dwie nowe metody molekularne do jednoczesnego wykrywania tych trzech gatunków nicieni, z których pierwsza umożliwia wykrywanie tych nicieni podczas jednej reakcji PCR i identyfikowanie ich na żelu agarozowym, a druga, bardziej czuła, umożliwia ich wykrywanie podczas jednej reakcji real-time PCR.
a. Multipleks PCR
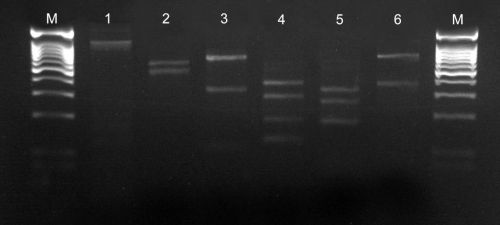
W metodzie tej opracowane zostały trzy specyficzne startery forward dla nicieni B. xylophilus, B. mucronatus i B. fraudulentus oraz jeden uniwersalny dla nich starter reverse. W wyniku przeprowadzonej reakcji PCR następowała skuteczna amplifikacja DNA dla tych trzech gatunków nicieni. Dla każdego badanego gatunku nicienia uzyskiwano na żelu agarozowym tylko jeden produkt, dla B. xylophilus otrzymywano prążek o wielkości 767 pz, dla B. mucronatus – 305 pz, a dla B. fraudulentus – 132 pz (Ryc. 19.). Specyficzne startery forward zaprojektowane dla trzech badanych gatunków nicieni powodowały otrzymywanie produktów PCR, wyraźnie różniących się pomiędzy sobą wielkościami, przez co były one łatwo odróżniane na żelu agarozowym. Specyficzność zaprojektowanych starterów testowana była na zgromadzonych populacjach B. xylophilus, B. mucronatus oraz B. fraudulentus, pochodzących z różnych rejonów świata (szczegóły metodyczne podane są w publikacji Filipiak i in., 2017). Wprowadzenie wszystkich trzech specyficznych starterów forward oraz startera uniwersalnego reverse do jednej mieszaniny reakcyjnej powodowało, że podczas reakcji PCR następowała amplifikacja produktu specyficznego tylko dla tego gatunku nicienia, którego DNA zostało wprowadzone do mieszaniny. W przypadku wprowadzenia do jednej mieszaniny reakcyjnej DNA jednego, dwóch lub wszystkich trzech DNA badanych nicieni, uzyskiwano na żelu produkty specyficzne zarówno dla B. xylophilus, B. mucronatus oraz B. fraudulentus. Pozwoliło to tym samym na jednoczesne wykrycie wszystkich trzech badanych gatunków nicieni podczas jednej reakcji PCR.
W celu potwierdzenia specyficzności opracowanych starterów, w badaniach wykorzystano również DNA innych gatunków nicieni występujących w sośnie, tj. B. piniperdae, B. pinophilus oraz Parasitaphelenchus papillatus, oraz DNA gatunku B. populi. Nicień ten występuje w topoli, jednak jest czwartym w Europie gatunkiem z grupy xylophilus (poza B. xylophilus, B. mucronatus oraz B. fraudulentus), wykazującym bliskie pokrewieństwo genetyczne i morfologiczne do kwarantannowego B. xylophilus. Dla żadnego z tych badanych gatunków nicieni, przy zastosowaniu zaprojektowanych starterów, nie otrzymywano na żelu agarozowym żadnych produktów PCR. Dla prób kontrolnych również nie otrzymywano na żelu żadnych produktów.
Przeprowadzone doświadczenia potwierdziły wysoką skuteczność zaprojektowanych starterów do wykrywania i identyfikacji podczas jednej reakcji PCR nicieni gatunków B. xylophilus, B. mucronatus oraz B. fraudulentus, mogących występować równocześnie w tych samych próbach drewna. Zarówno reakcje z DNA wyizolowanym z populacji nicieni, jak i z pojedynczych osobników (bez względu na stadium rozwojowe) pozwalały na wyraźne odróżnienie od siebie tych trzech gatunków nicieni. W porównaniu z innymi metodami molekularnymi opracowana technika multipleks PCR może być dużo prostszym i znacznie tańszym sposobem identyfikowania i odróżniania kwarantannowego szkodnika B. xylophilus od innych, niepatogenicznych gatunków nicieni występujących w drewnie sosny. Metoda ta pozwala również na znaczne zaoszczędzenie czasu, gdyż podczas jednej reakcji PCR możliwe jest jednoczesne zidentyfikowanie wszystkich trzech badanych gatunków nicieni.
Ryc. 19. Rozdział elektrofortetyczny dla reakcji multiplex PCR z zastosowaniem specyficznych starterów forward oraz uniwersalnego startera reverse. Gatunki nicieni testowane w badaniach: Bx1, Bx2 – B. xylophilus, Bm1, Bm2 – B. mucronatus, Bf1, Bf2 – B. fraudulentus, Bx + Bm – B. xylophilus + B. mucronatus, Bx + Bf – B. xylophilus + B. fraudulentus, Bm + Bf – B. mucronatus + B. fraudulentus, Bx + Bm + Bf – B. mucronatus + B. mucronatus + B. fraudulentus, Bp – B. populi, Bpini – B. piniperdae, Bpino – B. pinophilus, Pp – Parasitaphelenchus papillatus, H2O – kontrola odczynnikowa
b. Multipleks real-time PCR
W celu dalszego zwiększenia skuteczności procesu jednoczesnego wykrywania B. xylophilus, B. mucronatus i B. fraudulentus przeprowadziliśmy badania umożliwiające ich wykrywanie podczas reakcji multipleks real-time PCR. Metoda ta pozwala na zwiększenie czułości wykrywanych nicieni w próbach drewna.
W badaniach tych opracowane zostały dwa uniwersalne startery forward i reverse oraz trzy specyficzne sondy TaqMan dla nicieni B. xylophilus, B. mucronatus i B. fraudulentus. Reakcja PCR z zastosowaniem uniwersalnych starterów skutecznie amplifikowała produkty o wielkości około 270 pz dla wszystkich badanych nicieni. Specyficzność zaprojektowanych starterów testowana była również na zgromadzonych populacjach B. xylophilus, B. mucronatus oraz B. fraudulentus, pochodzących z różnych rejonów świata (szczegóły metodyczne podane są w publikacji Filipiak i in., 2019). W celu potwierdzenia specyficzności opracowanych starterów w badaniach wykorzystano również DNA innych, często występujących w sośnie gatunków nicieni, tj. B. piniperdae, B. pinophilus oraz Parasitaphelenchus papillatus, jak również DNA gatunku B. populi – czwartego, występującego w Europie gatunku z grupy xylophilus. Startery testowano w różnych warunkach oraz koncentracjach DNA.
Zaprojektowane startery uniwersalne i specyficzne sondy TagMan dla B. xylophilus, B. mucronatus oraz B. fraudulentus umożliwiły precyzyjne zidentyfikowanie tych gatunków i odróżnienie ich od siebie. Sonda zaprojektowana dla B. xylophilus amplifikowała tylko DNA badanych populacji tego gatunku. Przy zastosowaniu tej samej sondy dla DNA nicieni B. mucronatus oraz B. fraudulentus nie obserwowano żadnej amplifikacji. Analogiczne wyniki uzyskano dla reakcji z wykorzystaniem specyficznej sondy zaprojektowanej dla B. mucronatus oraz B. fraudulentus (Ryc. 20). Zarówno reakcje z DNA wyizolowanym z populacji nicieni, jak i z pojedynczych osobników (bez względu na stadium rozwojowe) pozwalały na jednoznaczne odróżnienie tych gatunków nicieni od siebie.
Dla innych badanych gatunków nicieni, tj. B. piniperdae, B. pinophilus, B. populi oraz P. papillatus, z zastosowaniem wspomnianych starterów i sond, nie otrzymywano żadnych produktów amplifikacji. Podobnie dla prób kontrolnych.
Ryc. 20. Krzywe amplifikacyjne dla reakcji real-time PCR. Do każdej reakcji wprowadzano trzy populacje gatunków Bursaphelenchus xylophilus, B. mucronatus oraz B. fraudulentus, uniwersalne startery forward i reverse oraz specyficzne sondy: A – sonda specyficzna dla gatunku B. xylophilus; B – sonda specyficzna dla gatunku B. mucronatus; C – sonda specyficzna dla gatunku B. fraudulentus (kolor czerwony – B. xylophilus, kolor niebieski – B. mucronatus, kolor zielony – B. fraudulentus, kolor popielaty – kontrola)
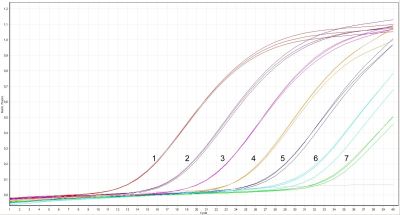
Czułość zaprojektowanej techniki analizowana była z wykorzystaniem serii 10-krotnych rozcieńczeń, począwszy od prób DNA B. xylophilus, B. mucronatus i B. fraudulentus o stężeniu 30 ng/µl. Przeprowadzone badania wykazały, że przy zastosowaniu opracowanych starterów i sond możliwe jest wykrywanie nicieni na podstawie nawet tak małej ilości DNA, jak 30 fg/µl (Ryc. 21).
Ryc. 21. Krzywe amplifikacyjne dla serii rozcieńczeń DNA z zastosowaniem uniwersalnych startów i specyficznych sond: A – seria rozcieńczeń DNA dla Bursaphelenchus xylophilus; B – seria rozcieńczeń DNA dla B. mucronatus; C – seria rozcieńczeń DNA dla B. fraudulentus (1–7 – seryjne rozcieńczenia DNA)
Przeprowadzone doświadczenia potwierdziły wysoką skuteczność zaprojektowanych starterów i sond TaqMan do identyfikacji i odróżniania B. xylophilus, B. mucronatus oraz B. fraudulentus podczas jednej reakcji. Podobnie jak w przypadku techniki multipleks PCR, opracowana technika multipleks real-time PCR również pozwala na jednoczesne wykrywanie tych trzech gatunków nicieni mogących równocześnie występować w tej samej próbie drewna. Niewątpliwą zaletą tej techniki jest jednak zdecydowanie większa czułość wykrywania nicieni. Ponadto metoda ta nie wymaga rozdziału elektroforetycznego uzyskiwanych produktów, co znacznie skraca czas otrzymywania wyników.
Opracowana technika multipleks real-time PCR została włączona przez Europejską Organizację Ochrony Roślin (EPPO) do protokołu diagnostycznego: „EPPO Bulletin (2022 w druku). Diagnostics PM 7/4 (4) Bursaphelenchus xylophilus, jako jeden ze standardów diagnostycznych obowiązujących we wszystkich krajach Europy do identyfikacji kwarantannowego nicienia węgorka sosnowca.
Projekt realizowany w ramach Dotacji Celowej MRIRW na 2026
Obszar 1: Ochrona roślin oraz ograniczanie zagrożeń związanych z rozprzestrzenianiem się organizmów kwarantannowych i stosowaniem
środków ochrony roślin.
Zadanie 1.3: Prowadzenie internetowej Platformy Sygnalizacji Agrofagów
Instytut Ochrony Roślin - Państwowy Instytut Badawczy
Ulica: Władysława Węgorka 20
60-318 Poznań
tel. +48 61 864 90 75
fax +48 61 864 91 20